 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 3 篇 doi: 10.5376/mpb.cn.2011.09.0003
收稿日期: 2010年11月28日 接受日期: 2010年12月15日 发表日期: 2011年01月19日
Susan R. McCouch & CGSNL (Committee on Gene Symbolization,Nomenclature and Linkage, Rice Genetics Cooperative), 2008, Gene Nomenclature System for Rice, Rice, 1(1):72-84 (doi: 10.1007/s12284-008-9004-9)
这篇文章全文,可在以下网址获得: http://www.springerlink.com/content/q047075330507t73/
水稻遗传协作组织的基因符号、命名法和连锁委员会(CGSNL, Committee on Gene Symbolization, Nomenclature and Linkage, Rice Genetics Cooperative)利用水稻全基因组测序完成和生物学界涌现出的新基因检测、验证和描述方法等有利条件,修订了水稻的基因命名法系统。本文概述了以被注释和定位在测序基因组组件上的DNA、RNA和蛋白序列信息,及生化特性和基于前向遗传学的表型特征为基础描述基因的标准程序。 通过这些修订,我们增强了物种间的结构、功能和进化比较的可能性,并努力使水稻的基因命名法系统和其他模式物种的保持一种和谐关系。 新鉴定的水稻基因现在可以在如下地址在线注册:http://shigen.lab.nig.ac.jp/rice/oryzabase_submission/gene_nomenclature/
生物学界一直梦想有一套统一的基因命名系统。目前已经有多个基因命名法系统来描述:拟南芥(TAIR, 2005)、番茄(http://www.sgn.cornell.edu/documents/solanaceae- project/docs/tomato-standards.pdf)、玉米(http: //www.maizegdb.org/maize_nomenclature.php)、苜蓿(VandenBosch and Frugoli, 2001)、酵母(http://www.yeastgenome.org/gene_guidelines.shtml)、小鼠(MGNC, 2005, http://www.informatics.jax.org/mgihome/nomen/)和人类(Wain et al., 2004)。一种跨越不同物种间通用遗传语言的使用将是科学界的一个巨大进步,将极大的便利于物种间的基因和遗传变异的结构、功能和进化比较。随着对基因 和基因产物分子和生化特性的重视,建立一套能反映特定基因、基因模型或基因家族生化特点和在特定遗传背景下一个特定等位基因的表型结果的水稻基因命名法系 统,显然十分重要。
目前水稻基因名称和基因符号规则是基于水稻遗传协作组织(CGSNL)中的基因符号、命名法和连锁委员会 (Kinoshita, 1986)的建议制定的。绝大多数早期的基因命名和符号是对可见表型的描述,为该基因的存在提供最早的证据,这些基因名称和符号被水稻研究领域普遍使用。 随着水稻全基因组测序的完成(IRGSP, 2005),和新的鉴定、定义和描述基因的新方法不断涌现,概述一套描述基因的标准程序的命名法系统成为了迫切需要,描述是以生化特性和DNA,RNA和 蛋白序列分析(Wu et al., 1991),及以前规范基因和表型联系的规则(Kinoshita, 1986)为基础。
本文集中总结了水稻的基因命名法规则,尽一切可能来在水稻基因命名法系统和其他模式物种的之间协调一致。本文种描述了一套染色体命名和基于生物学功能、突变表型和 序列鉴定座位、基因和等位基因的规则,并就如何处理不同来源的多种基因组组装注释种中的别名(同义名)、序列差异和座位提出了建议。这个命名法规则是以原 有的水稻基因命名法系统(Kinoshita, 1986)为基础的,但新的命名法系统被扩展来吸收国际水稻基因组测序项目(International Rice GenomeSequencing Project, IRGSP)成员在两次水稻注释项目(RAP)会议(RAP-1, 2004年12月, 日本, 筑波; RAP-2, 2005年12月, 菲律宾, 马尼拉)总结的有关序列信息的建议。这些规则也已经获得了水稻遗传协作组织中CGSNL小组委员会的讨论通过(http: //www.shigen.nig.ac.jp/rice/oryzabase/rgn/office.jsp)。
尽管有记录以来水稻遗传研究已经有超过一个世纪的时间,但最近在籼稻(Oryza sativa ssp.indica)和粳稻(Oryza sativa ssp.japonica) 大规模诱变实验和EST测序方面取得的进展,大大的增加了我们对基因网络、基因功能、等位基因和序列多态性的认识。因此,本次报告中概述的命名法主要是归 纳出以生物功能为基础的基因和等位基因的命名规则,方便多个测序和注释项目中基因注释的交叉参考,这些项目包括:国际水稻基因组测序计划(IRGSP) (IRGSP, 2005)、水稻注释计划(RAP) (Ohyanagi et al., 2006)、美国基因组研究所(TIGR) (Yuan et al., 2005)、慕尼黑蛋白质序列信息中心(MIPS) (Karlowski et al., 2003)、美国国家生物技术信息中心(NCBI) (http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?taxid=4530)、先正达 (Syngenta) (Goff et al., 2002)和北京基因组研究所(BGI) (Zhao et al., 2004),同时这将为来自不同种质资源测序(Ammiraju et al., 2006; McNally et al., 2006)中存在的基因差异体的注释提供内在连贯性。
1基因组组装和系统座位标识码(systematic_locus_ID)
一个简单的水稻物种也许可以支撑多重的遗传、物理、序列图谱、基因注释和基因组组装。目前水稻(O.sativa) 基因组分别被粳稻栽培种Nipponbare基因组序列(IRGSP测序)和籼稻栽培种93-11基因组序列(BGI测序)所代表。Nipponbare 的序列已经被几个研究小组进行了注释,包括了RAP (Itoh et al., 2007)、(Ohyanagi et al., 2006)、TIGR (Yuan et al., 2003)、NCBI-GenBank (http://www.ncbi.nlm.nih.gov/mapview/map_search.cgi?taxid=4530)、MIPS (Karlowski et al., 2003)和先正达(Goff et al., 2002),然而栽培种93-11的序列注释工作几乎全部来自BGI (Zhao et al., 2004)。对Nipponbare来说,IRGSP测序得到的原始序列数据被来RAP和TIGR各自独立组装和注释,因此水稻研究界目前管理着三个独立 的基因组组装(两个来自栽培种Nipponbare和一个来自栽培种93-11)。
这些组装代表着各自一套相互之间独立并有细微差别的对 座位的注释,这些座位代表着沿假设分子锚定排列的基因模型/转录单元。一个座位定义为基因组上的一个位置,因为每个注释小组都独立的依据在假设分子上的位 置,分配座位标识符(locus IDs)给所有的基因、转录本和蛋白。相同的基因可能因基因组、组装和注释软件的不同而赋予了不同的系统座位ID (systematic_locus_ID)。每个注释小组使用的为核基因/转录本/蛋白、细胞器基因/转录本/蛋白和转座本分配的系统座位IDs规则, 在RAP数据库(Ohyanagi et al., 2006)、TIGR Osa1数据库(Yuan et al., 2005)和BGI-RIS (Zhao et al., 2004)中有具体描述。来自(RAP)数据库的有关分配系统座位(IDs)的引证例子相关建议在本文的结尾部分有列举。
注释的基因包括了蛋白质编码基因(open reading frames, ORFs/CDSs)、非编码RNA基因(ribosomal RNA (rRNA), 转移RNA (tRNA), 微RNA, 小干扰RNA (siRNA), 小核RNA (snoRNA)等)和假基因组。系统座位IDs的使用(将在本文后部详细描述)为基因标识符的分配提供了一个系统的方法,同时也为座位在已测序水稻基因 组的位置提供了容易的识别。作为结果,座位ID可以被用来鉴定和在一个特定的基因组组装中追踪一个座位,在一个基因模型和功能注释基因之间建立关联。目前 大多数的序列和注释基因是未知功能(实验确认)的,系统座位ID也为跟踪这些假基因的功能提供了一个有用的办法。如在表1总结的,基因可以根据计算机鉴别 的序列与已知基因(推测的同源基因, 直系同源基因或旁系同源基因)、蛋白或共有序列的特征(像某蛋白的功能结构域)的相似性来进行分类。当序列的相似性不足以保证基因名的分配时,对基因特征 的记述信息就做出了关键的贡献。尽管系统座位标识符在一个基因组组装和注释数据集种提供独有的命名法,但不同注释小组使用的分配座位IDs的方法有细微的 差别,加上基因组组装和基因指令表的差别(亦即籼稻和粳稻),使得不同基因组组装版本之间最终的基因和座位交叉参考变得十分困难。因此,随着基因功能或表 型的实验证实和描述,CGSNL就提供了一个统一的独立于不同基因组组装和注释版本之外的基因追踪系统。正如下边描述的,每一个在CGSNL注册的基因都 可以通过基因的全称和一个基因符号获得独一无二的鉴别。
.png) 表1 CGSNL建议的已测序基因分类规则 Table 1 Rules for Classifying Sequenced Genes as Suggested by the CGSNL |
在CGSNL数据库登记基因将有助于多个注释系统,及等位基因和 序列变异体之间的基因的交叉参考。审订过基因名和符号的基因将和一个基因功能或表型相关联,在一切可能的情况下,在登记的时间研究人员被要求为来自RAP 注释数据库的新基因标示一个系统座位标识符。在任何可能的情况下,为系统座位IDs在其他注释数据库(即TIGR, BGI等)建立联系。因此,在注册登记的时间,当一个系统座位标识符被提供给CGSNL的时间,组装和注释的版本也必须被储存以提供全面的细致的相关档 案。如果可能,GenBank或DDBJ登录号也应该被提供。这将有助于确保在表2描述的交叉参考信息的正确和适用性。
 表2 SD1基因的例子及其相关信息 Table 2 Example of the SD1 Gene and Its Associations |
水稻假分子之间的交叉索引需要人工认真的选择和整理。就旁系同源基因,特别是当串联排列时,基因模型结构中在多套相同基因组序列组装版本之间存在的细微差别都会带 来巨大的挑战。如果研究人员对一个新基因的这些特定性状谙熟,自己将处于最有利的位置为提供这个基因准确信息方面,这将确保不同水稻基因组注释差别之间的 渐进改进和更新。
2水稻中染色体名和基因符号命名规则
应这个命名法系统的需要,一个基因被定义为一段有已知或预测功能或表型的DNA序列。那些测序的基因如果没有实验证明的明确功能或表型,CGSNL将不会分 配其一个基因名或基因符号(图1)。那些功能或表型已经被经典遗传学确认的基因,但如果没有和其关联的序列,将分配给一个基因名和基因符号,但也许不会分 配给一个系统座位ID。
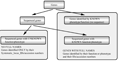 图1 基因赋予全称的图示"基因"是指水稻的全套基因 Figure 1 Schematic representation of genes receiving full names |
2.1染色体名称
以Khush 和Kinoshita (Khush and Kinoshita, 1991)提出的约定为基础,水稻的12条核染色体赋予了阿拉伯数字,连锁群与染色体建立了对应关系,并得到命名。为方便数据库建设的目的,每个染色体被 赋予了01到12的一个两位数字符,但1到9的单位数字经常在出版物中出现。断臂和长臂分别标示为“S”和“L” (例如: 1S, 1L),因此chr.1S和chr.1L或Chr.2S和Chr.2L的缩写是被接受的。尽管由于当时评估染色体大学和臂比技术的准确性不够,染色体和染 色体臂命名的约定目前公认有不一致性,但本次并没有对存在的染色体命名法提供修订建议。环状的线粒体或质体和叶绿体染色体分别分配了英文符号“Mt”和 “Pt”,而没有使用类似核染色体中的阿拉伯数字。这些不具有着丝粒的染色体将不被指定短臂或长臂。这些染色体可以缩写为chr.Pt或Chr.Mt。
2.2基因全称
一个基因全名包括了基因名和这个基因座的标识符编号。基因名全称所有字母大写斜体,在名称和座位编号之间有一个空格(即, SHATTERING 1)。 这个基因名应该简要的描述与基因产物的生化功能或由于突变或基因的等位基因形式所呈现出的表型所关联的显著特征。基因座位编号由1到3位数的数字组成,用 来区分一个特定基因座位上的基因与其他座位上基因的具有相似的功能和表型。座位标志符使用的数字表明一个特定基因或基因家族的被鉴定的次序,不应该和系统 座位ID或其所在的染色体/连锁群混淆。默认的情况是,任何基因名如何没有一个座位标志符,就代表这个基因是第一被鉴定的此类基因,将分配给座位标志符 “1”,例如,PURPLE NODE就被标示为PURPLE NODE 1。基因全称的写法是全部大写、斜体,这和先前的规则:基因全称第一个字母大写代表第一个等位基因显性,小写代表隐性;其他所有的其他字母小写、斜体。有关此的更多内容,请参考“显性/隐性关系”章节。
当一个表型定位到一个复杂座位,这个座位包含一个串联的基因家族(例如, XANTHOMONAS ORYZAE PV. ORYZAE RESISTANCE 21, XA21,或者SUBMERGENCE 1, SUB1)的情况下,这个串联排列基因家族中的每个基因都会被分配给一个独立的座位标识符(即SUB1, SUB2, SUB3等)。
如有一个基因是通过序列信息得到验证的,这个基因在后边被证明和一个基于表型验证的基因相同(如Kinoshita (1986)列出的那些),那么这个基因 全称和规则的施用将基于表型,赋予其他名称作为同义名。如果不同的基因使用同样的名或同一个基因使用不同名称造成重复、冗余或混淆,第一个公开出版的基因 名将被保留,CSGNL将和出版发布这个基因的作者联系,赋予这个基因一个新的基因名和基因符号来用于以后对这个基因或座位的使用。质体和叶绿体基因组中 鉴定的基因将按照Uniprot描述的那样分配名称和符号,线粒体基因组中的基因将按照推荐的规则分配名称和符号(http: //ca.expasy.org/cgi-bin/lists?plastid.txt)。
基因的名称是基于实验验证的基因功能或表型效 应来分配的。实验证据也许表明了一个分子功能、生物途径中的作用,与另一个基因的相互作用,或与这个基因有联系的表型(图1)。那些基于计算机验证的与同 源物、直系同源物、旁系同源物、或一个保守区域如Interpro结构域(Mulder et al., 2005)的序列相似性的基因,只有在有充分的实验证据确认基因功能时才会分配给基因名。2004年11月在日本筑波市举办的水稻注释计划(RAP-1) 会议的参会人员同意,数据库的管理人员可以使用一个标准的“证据分类”系统来表明证据的类型或出版提供的有关核基因注释的实验证据。有关这些分类的描述可 以在表1找到。如CGSNL规定:如果证据被认为不足以证明分配的基因功能,这个基因名字段将保留空白,描述/定义字段将被用来对这个基因特征的已知内容 的进行描述(表1)。
2.3基因符号
基因符号是基因全称的缩写,用斜体表示。一个基因符号包括两个部分,即基因的分类符号包括2到5个字母,和对应的座位标识符包括1到3位字符。基因符号衍生于先 前讨论的基因名全称,因此和基因名全称一样使用相同的座位标识符。基因符号的两部分应该写到一起,中间不留空格、连字符或其他符号(例: SH1, GLH2)。基因分类号和座位标识符一起组成了基因符号,必须对这个座位和基因组来说是唯一的。基因符号的分配原则就是容易和一个基因名全称对应并辨识。在任何地方,如何存在的标识符如果不能完全符合这个规则,就应该被保留,例如:C (CHROMOGEN FOR ANTHOCYANIN),A (ANTHOCYANIN ACTIVATOR)和WX (GLUTINOUS ENDOSPERM)。对任何没有座位标识符的基因符号来说,会被默认为座位标识符为“1”,例如,GLUTINOUS ENDOSPERM (WX)应该指定为GLUTINOUS ENDOSPERM 1(WX1)。所有具有相似特性的新基因将被CGSNL根据发现的顺序分配给一个新的座位标识符。CSGNL将保证先前鉴证的基因符号和新鉴定登记的基因分配到一个唯一的基因符号,避免名称和符号的混淆。
使用后缀“(t)”和“*”来表明一个假设性的座位标识(当一个新的描述基因和一个先前已知基因的等位基因关系不是很清楚(Kinoshita, 1986))被暂时使用,在假定其为新的座位的情况下,新基因将被分配给一个新的座位标识符。如果这个新基因在以后被证明和原来已知座位是等位的,两个相 关记录将被合并,最初的基因符号将被按照程序规则采纳。其他符号将会作为同义词引用。以前分配的基因符号将不会删除,这可以避免相同符号重新使用导致的混 乱。分配一个符号给一个基因的时候应像上边描述的那样,保持和基因名全称的一致性。
作者在其文章种涉及到已知功能的水稻基因的时间,一定要引用核 准的基因名全称和符号,如果有可能要引用基因组注释中心之一的系统座位ID和GenBank登录号。当完整的信息不存在的情况下,除非有额外的实验证据的 提供,否则系统座位ID或基因符号将不会被使用。只有通过CGSNL的审查,基因名才可以被分配使用。
3物种名在基因名和符号中的使用
出版物中在基因名和基因符号前使用物种特异性前缀如“Os” (O.sativa)也许是有用的,但这并不在官方已经命名规则中,因为对已经和物种信息关系的递交/注册基因来讲显得有些冗余。而且,也会导致基因名Oryza sativa-X的 扩散。基因和物种之间的关系会在所有的基因组数据库和学历数据库中清楚的保留。然而,作者可以在出版物中附加上物种特殊性的前缀,以此来避免在任何时候参 考一个基因时所带来的物种名重复。在任何情况下,物种符号都不应该变成被采用的基因符号或基因名全称的一部分。特别需要指出的是,符号“Os”在系统座位 ID中是允许使用的,例如,Os05g0000530、LOC_Os03g01590和OsIBCD000082这些分别为RAP (http://rapdb.lab.nig.ac.jp/index.html)、TIGR (http://www.tigr.org/tdb/e2k1/osa1/tigr_gene_nomenclature.shtml)和BGI-RIS (http://rise.genomics.org.cn/rice/index2.jsp)数据库所采用。
3.1等位基因变异体
同一个基因的不同的等位基因是通过添加数字后缀来区分的,数字和基因全称或基因符号之间有破折号或连字符分开,例如,SHATTERING 1-1(SH1-1),PGI1-1和PGI1-2。在历史上,有几种情况下曾用过字母(t)或星号(*)而不是数字来指明一个等位基因,因为这些字母符 号在描述等位基因变异体时广泛使用并被水稻遗传研究学界接受,这些字符将在出版物中作为例外保留,并将在数据库中作为同义名标明。
3.2显性/隐性关系
历史上,基因全称是全部小写斜体,如果文献中第一个等位基因是显性的就以一个大写字母开头,如是隐性就以小写字母开头。由近来在大规模基因组测序努力为基础 在基因鉴定取得的进展,那些仅仅在单倍体细胞(即花粉或卵)中表达基因,一个等位基因的显性或隐性或一个座位的变异体,也许就是未知的或无关紧要的。然 而,在调查基因功能的时候,一个等位基因的显/隐性对遗传研究来说依然是十分重要的。因此,为出版的目的,建议每一个特定的种质资源被描述的地方,隐性等 位基因都使用小写字母,显性等位基因第一个字母大写,其他全部小写,这两种情况下所有字母都是斜体(如先前的约定; 表3)。但正式的(全称)基因名称所有字母需大写,特定等位基因的显隐性表型将作为这个等位基因的属性,而不是在数据库中基因名称的一部分被记录。
 表3 在文献中基因名全称和符号使用的例子 Table 3 Example of a Gene Full Name and Symbol for Use in Publications |
3.3序列变异体
考虑到一个基因就是一段具有已知或预测功能或表型的DNA片段,一旦一个基因通过一个系统座位ID被命名并定位到一个序列图谱上,它就可以被一群定位在同一 个遗传座位的等位基因和序列变异体表示。在不同植物材料中仅仅通过序列鉴定的分子序列变异体将被赋予一个名称、符号和登录标识码,有关这个序列变异体的相 关信息将交叉参考形成有关这个种质资源(包括对应的种质登记ID)的特定信息,从这些种质中DNA/RNA材料被提取。然而,序列变异体将不会被 CGSNL认为是“等位基因”,除非它们具有分子功能或有描述的表型,并且经过等位性试验的证明。那些特定功能未知的“序列变异体”将通过添加给一个等位 基因名后缀“-sX”来区分“等位基因”,“s”表示“已测序”,“X”是鉴别一个特定序列变异体的编码。一个分子变异体的名称和符号获得一个对应基因的 名称和符号,这和一个等位基因的约定相似,除了其带后缀的描述,并且由于不能分配给这些测序变异体等位表现而全部大写(表3)。
如果一个 测序的变异体以后被证据并赋予一个新的特定表型或功能,它将被分配给一个新的等位基因标识符,或如果一个测序变异体被证明和一个先前命名的等位基因对应一 个已知基因相同,它将被基于程序规则分配给一个存在的等位基因标识符,原来其他标识符将作为同义名保留。一个推荐的基因座位、全称和等位基因命名的例子在 表3。序列变异体鉴定相关的种质名和其登记号信息将不会在正式的名称和符号中记录。这部分信息将单独记录在数据库中,以方便遗传学界的交叉参考。提交序列 变异体的作者有责任来搜寻这个新的序列形式是否和以前报告的序列变异体或等位基因相同。在文献中,作者可以选择等位基因名、序列变异体后缀和种质资源串联 起来,避免对读者来说过度的重复。
3.4蛋白质名和符号
一个特定基因编码的蛋白,其名称在该基因名称是基于表型或分子功能的情况下(参考“基因全称”章节),应该和这个基因的全称保持一致,但蛋白的名称将全部大 写并且不需要斜体。如果在后期阶段,一个基因和它对应的蛋白产物被证明有一个生化分子功能,如是一个酶或一个大分子复合物的结构组成部分(亚基),这个蛋 白应该按照IUPAC酶学委员会建议的酶命名法或IUBMB的大分子命名法分配一个同义名(Committees Biochemical Nomenclature, 2006)。对于一个特定的蛋白来说可能会有多个功能分配(即, 基于表型试验, 生化分析或分子功能),因此一个蛋白名会有几个异名(和基因名全称相似)。蛋白符号应该一直和采纳的基因符号保持一致,除了蛋白符号全部大写不用斜体,紧 接一个空格和数字的座位标识符。例如,GLUTINOUS ENDOSPERM 1(WX1)基因编码谷粒表面淀粉合成酶(EC: 2.4.1.11)。蛋白名是GLUTINOUS ENDOSPERM 1和符号“WX1”。蛋白名‘WAXY’、‘WAXY 1’和GRANULE-BOUND STARCH SYNTHASE (GBSS)将被当作同义名记录。如果一个名称不能基于表型、已知生化或其他实验证据支持其他功能而得到分配,一个系统座位标识符(上述)和一个和表1描 述一致的名称必须用来描述这个基因,直到其功能得到确认。
3.5转录后修饰
当存在转录后修饰时,如蛋白质剪接导致两个或更多具有不同活性或功能的蛋白质分子的形成,剪接蛋白分子将获得和它们分子功能或关联表型一致的蛋白质名称和符号,初始分子的名称和符号作为同义名。
3.6假基因
分子技术鉴定了和结构基因序列十分相似的相同序列,但不被转录,这些序列称为假基因。为了表明这些假基因和功能基因的相关性,假基因将被使用和结构/功能基 因相同的基因符号,名称斜体表示,后边加上字母“.P”(符号“.”和大写字母“P”)。这将取代通常使用的希腊符号“psi”作为假基因的标记;一个例 子是RPS14.P取代RPS14.psi作为假核糖体蛋白S14的符号。同样的命名法建议用于线粒体和质体(叶绿体)基因组的假基因,例子就是 ACTB.P1 (ACTIN BETA PSEUDOGENE 1)、ACTB.P2 (ACTIN BETA PSEUDOGENE 2)等。假基因可能在不同的染色体上或者和功能基因紧密靠近,据此得到它们的名称并存在变化的编号。从命名法的角度来看,一个假基因就是一个没有功能的基 因(http://pseudogene.org/main.html.)。如果一个假基因以后被证明可以转录并调控另外一个基因的表达或转录的mRNA 具有一定功能,这个基因必须重新划归另一个基因类别如fnRNA或potogene (Brosius and Gould, 1992)。
3.7未定位基因
由于一个物种中固有的遗传变异性,来自某一种质的一个基因有可能在水稻两个已测序全基因组中,由于插入/缺失多态性和基因家族的扩增/收缩而不能定位。同 样,在分离群体中通过表型鉴定出的一个基因也可能不存在于两个双亲的基因组中。在这种情况下,即使没有定位信息,基因名称和符号也应该分配给这些等位基因 变异体。当给一个未定位座位分配基因名称时,必须有有效的实验证据支持这个基因和其功能的存在。如有一个确认相似未定位测序基因第二审存在的话,最好的相 互匹配的方法应该是更加严格的确认是否事实上它和先前鉴定的基因是同样的。在二审表现型确认基因存在的情况下,等位性或互补试验作为必需证据被考虑进去。 如有任何这些证据的缺失,这个基因就将被分配一个新的基因名称和符号。同时,唯一标识符将被分配给CGSNL登记的这个基因,如果有Genbank登录号 存在的话,将作为占位符。
3.8数量性状遗传座位(QTL)
QTLs座位基因的位置标识符对基因组功能特征有贡献。一个QTL代表一个统计学上的可测表型,概括来说就是一个数量遗传性状。通过分离群体的连锁遗传作图鉴定的QTLs,每一个QTL被定义为至少通过两个紧密连锁的定位遗传标记限定的染色体上特定区间。
水稻QTL命名法规则(McCouch et al., 1997)指出,每一个QTL名称都要以小写斜体字母“q”来表明是其是一个QTL,紧跟着就是2到5个字母的标准“性状名称”(如, SW标示粒宽),一个数字表明其所在的水稻染色体(1-12),一个“.”和一个区别于其他在同一个染色体上的单个QTLs的唯一标识符(例如, qSW5.1)当QTLs进入一个基因组数据库例如Gramene (Jaiswal et al., 2006),它们将被分配给一个来自与性状主体的标准性状术语(TO; 例如, 籽粒宽度, 登录号#TO: 0000140) (Jaiswal et al., 2002)来方便查询,也许可能分配一个新的唯一标识符来避免研究中间的混乱。在任何情况下,这个数据库赋值将作为这个QTL记录中的一个同义名,原始的 文献中的QTL名称将为检索的目的而得到保留。
当这些负责具体表型变异的QTL就是这些基因,并且第一次基于对应的QTL而被鉴别出来 时,基因名称全称可以反映QTL的标示(除了除掉前缀‘q’外, 还要使用斜体(例如, SW5));然而,如果对应QTL的基因和一个先前命名和描述的基因想符合,流程规则的使用和原始基因的名称必须被保留。但是,建议基因和QTLs之间的 关系要在和基因名关联的同义名列表中注明。
4系统座位ID分配:一个RAP数据库例子
4.1核基因的系统座位ID
系统座位标识符将依次分配给水稻(O. sativa ssp. japonica, cv. Nipponbare)假分子(水稻测序基因组组装染色体重叠群)基于自动基因预测程序、直系同源联配、和/或ESTs和全长cDNAs联配鉴定出的基 因,遵循酵母(S.cereviseae) (http://www.yeastgenome.org/gene_guidelines.shtml)和拟南芥(A.thaliana) (TAIR, 2005)。系统标识符被分配给蛋白编码基因(ORFs)、RNA编码基因(snoRNA, snRNA, rRNA, tRNAs, and microRNAs)、和假基因。一个核基因座位ID将包括:(a)一个大写字母“O”和小写字母“s”来标明水稻物种O .sativa;(b)两个位数的数字表明特定的水稻染色体(01, 02, 03, …12);(c)一个字母“g”来标明这个座位ID是一个基因;(d)一个7位数数字(假设每个染色体上有不少于10 000个基因)标明基因在染色体上的顺序,按照从端粒的短臂(北端)到端粒的长臂(南端)的升序排列。标明基因顺序的数字是独立于染色体链的极性的(+/ -或Watson/Crick),并且起始的时间就分配了100的增量,为新基因的发现扩增留下空间。例如,染色体5上的第三个和第四个基因被标示为 Os05g0000300和Os05g0000400。
在测序过程中或有新的实验证据表明一个新基因在两个基因注释的基因间被辨别出来, 这个新基因将被使用后边第十位数字位置,分配一个两个先前已的注释基因之间的一个数字。例如,在基因Os05g0000300和Os05g0000400 发现的基因可以分配给Os05g0000350,而且留下扩增的空间。尽管这个策略有很明显的优势,但在一些情况下在一个特定的染色体片段中基因的顺序并 没有遵循基于基因发现优先顺序的升序/降序规则;然而,这些缺